[충청뉴스 이성현 기자] 국내 연구진이 내성 암세포를 다시 약물에 반응하게 만들 수 있는 핵심 유전자를 자동으로 예측하는 컴퓨터 기반 방법론을 개발했다.
한국과학기술원(KAIST)는 생명화학공학과 김현욱·김유식 교수 연구팀이 인체 대사를 시뮬레이션할 수 있는 컴퓨터 모델인 대사 네트워크 모델을 활용해 항암제에 내성을 가진 유방암 세포를 약물에 민감화시킬 수 있는 새로운 약물 표적을 예측하는 컴퓨터 기반 방법론을 개발했다고 7일 밝혔다.
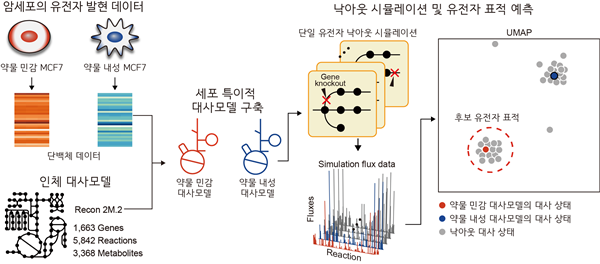
연구진은 암세포의 대사 변형이 약물 내성 형성에 관여하는 주요한 특징으로 주목하고 항암제 내성 유방암 세포의 대사를 조절해 약물 반응성을 높일 유전자 표적을 예측하는 대사 네트워크 모델 기반 방법론을 개발했다.
연구진은 먼저 독소루비신(doxorubicin)과 파클리탁셀(paclitaxel)에 각각 내성을 지닌 MCF7 유방암 세포주에서 얻은 단백체 데이터를 통합해 세포별 대사 네트워크 모델을 구축했다. 이어 모든 대사 유전자에 대해서 유전자 낙아웃(결실) 시뮬레이션을 수행하고 그 결과를 분석했다.
그 결과 특정 유전자의 단백질을 억제하면 항암제에 잘 듣지 않던 내성 암세포가 다시 항암제에 반응하도록 만들 수 있다는 것을 알아냈다.
독소루비신 내성 세포에서는 GOT1 유전자를, 파클리탁셀 내성 세포에서는 GPI 유전자를 선별했으며, 두 약물 공통으로는 SLC1A5 유전자를 표적으로 선별했다.
선별한 유전자를 실제로 억제해 본 결과 내성 암세포가 항암제에 다시 반응하게 됨을 실험적으로 검증했다.
나아가 같은 항암제에 내성을 갖는 다른 종류의 유방암 세포에서도 같은 유전자를 억제했을 때 항암제에 다시 민감해지는 효과가 일관되게 나타나는 것을 확인할 수 있었다.
김유식 교수는 “세포 대사는 감염병, 퇴행성 질환 등 다양한 난치성 질환에서 중요한 역할을 한다”며 “이번에 개발된 대사 조절 스위치 예측 기술은 약물 내성 유방암 치료를 넘어, 치료제가 없는 다양한 대사 질환에도 적용될 수 있는 기반 기술이 될 것”이라고 설명했다.
김현욱 교수는 “이번 연구의 가장 큰 의의는 컴퓨터 시뮬레이션만과 최소한의 실험 데이터만으로 내성 암세포를 다시 약물에 반응하게 만들 수 있는 핵심 유전자를 정밀하게 예측할 수 있다는 점”이라며 “이 방법론은 다양한 암종과 대사 관련 난치성 질환의 새로운 치료 표적 발굴에도 폭넓게 활용될 수 있을 것”이라고 말했다.


